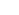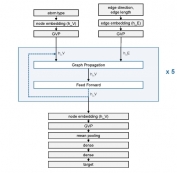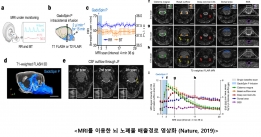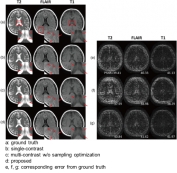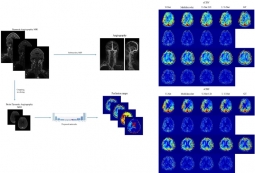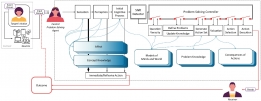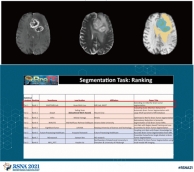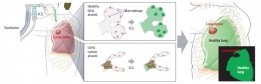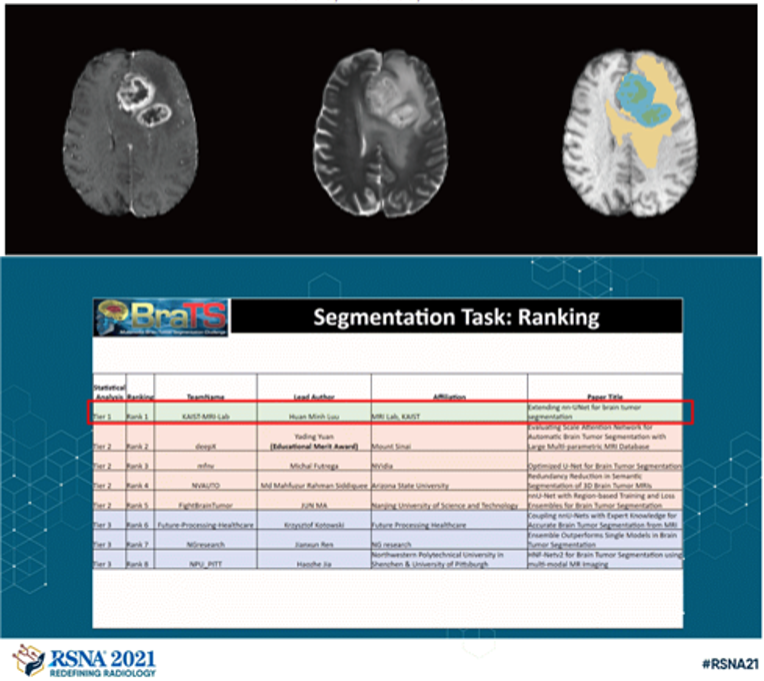
<본 연구팀이 제안한 뇌종양 구획화 모델 대표 이미지(위), BraTS 대회 딥러닝 모델성능 순위(아래)>
이번 달에는 최근 MICCAI/ASNR 주최의 brain tumor AI challenge 2021 (BraTS 2021)에서 해외유수의 AI 연구팀들과 경쟁하여 1위를 차지한 박성홍 교수님 연구실의 Luu Minh-Huan 박사과정 학생과 인터뷰를 진행해보았습니다.
Q1. 바쁘신 와중에 인터뷰에 응해주셔서 감사합니다. 우선 자기소개 부탁드립니다.
안녕하세요, 저는 Luu Minh Huan이고 박성홍 교수님 MRI 연구실 박사과정 3학년 학생입니다. 저는 베트남에서 온 유학생이며, 학부때부터 바이오 및 뇌공학과를 다녔습니다. 현재 주요 연구분야는 의료 영상을 위한 딥러닝 개발과 정량적 MR 영상기법 개발을 진행하고 있습니다.
Hello, my name is Luu Minh Huan and I’m a third year Ph.D student in professor Park Sung Hong’s MRI laboratory. I am an international student from Vietnam and I have been in Bio and Brain Engineering department since my bachelor degree. My current research involves deep learning applications for medical imaging as well as developing quantitative MR imaging methods.
Q2. Brain tumor AI challenge는 어떤 것인지 설명해주실 수 있을까요? 어떤 계기로 참여하게 되셨나요?
뇌종양 구역 분할 대회인 BraTS는 2012년에 시작된 국제 대회입니다. 이 대회에서 참가자들은 BraTS 주최측에서 제공하는 고품질의 T1, 조영 증강, T1/T2 및 FLAIR의 4가지 다른 MRI 영상에서 뇌종양 영역을 분할하는 딥러닝 알고리즘을 개발하고 그 성능으로 경쟁합니다. 이 대회는 주로 MICCAI 커뮤니티가 주관해왔지만 10회째인 올해는 MICCAI, RSNA(북미 방사선학), ASNR(미국 신경방사선학회) 등 3개 기관이 팀을 이뤄 기존 대회보다 훨씬 더 많은 데이터를 제공하여, 이번 대회는 엔비디아 등 주요 AI업체를 비롯한 여러 나라와 연구기관에서 참가했습니다.
뇌종양의 정확한 분할은 치료 계획 또는 치료 진행 모니터링 등 임상활용적 측면에서 매우 중요합니다. 그러나 수동으로 뇌종양 영역을 구획화할 경우 매우 지루하고 시간이 많이 걸리며 전문지식이 필요합니다. 딥러닝 알고리즘을 통해 이 과정을 가속화하고 치료 과정을 간소화할 수 있습니다. 제가 BraTS에 참여하게 된 주된 동기는 딥러닝 분야를 조금 더 직접 배우고 싶기 때문이었습니다. 뇌종양 영역분할은 특히 더 활발한 연구분야로 새로운 딥 러닝 기반 모델이 자주 제안되는 분야입니다. 이런 대회에 참가함으로써, 저는 저나 다른 참가자들에 의해 개발된 많은 다른 모델들의 성능을 공정하고 편견 없는 평가 시스템과 직접적으로 비교할 수 있었고, 데이터 전처리 방법과 뇌 데이터를 위한 딥러닝 훈련을 설정하는 방법등을 배울 수 있었습니다.
BraTS, short for brain tumor segmentation challenge, is an international competition that started in 2012. In this competition, participants develop automatic algorithms to segment the tumor sub-regions from 4 MRI contrasts: T1, contrast-enhanced T1, T2, and FLAIR (fluid attenuated inversion recovery). Accurate segmentation of the brain tumor is critical for the many clinical applications, such as treatment planning, image-guided interventions or monitoring of treatment progress, etc. However, manual segmentation is very tedious, time-consuming, and requires expertise. With automatic algorithms, we can accelerate this process and streamline the treatment process. The BraTS competition provides high-quality clinical scans, allowing researchers to develop and compare algorithms on a common benchmark. The competition is usually organized by the MICCAI community but for this year, which is the 10th competition, three organizations, MICCAI, RSNA (north America radiology), and ASNR (American society of neuroradiology) teamed up and provided significantly more data than previous competitions. Participants to this competition come from many countries and research institutions, including leading AI companies such as NVIDIA.
My main motivation for participating in the BraTS competition is to learn more about the field. Brain tumor segmentation is an active area of research, especially with many new deep learning-based models being proposed frequently. By participating in the competition, I can directly compare the performance of many different models, developed by me or other participants, with a fair and unbiased evaluation system. I also learned how to preprocess the data and setting up trainings for brain data.
Q3. 이번에 BRATS brain tumor segmentation challenge에 발표하신 모델의 특징과 강점이 무엇이며, 이를 구현하기 위해 어떤 개발을 진행하셨는지 여쭤보고 싶습니다.
저의 주된 접근 방식은 작년 우승자의 딥러닝 모델을 개선하는 것입니다. 다행히도 기존 개발자분들께서 모델 개발에 필요한 코드와 개발 과정을 잘 정리하여 제공해주셔서 도움이 많이 됐던 것 같습니다. 작년 모델을 밑바탕으로 2가지 주요 개선사항을 적용하였습니다. 첫번째는 올해 주최측에서 제공한 데이터의 크기가 4배 더 크기 때문에 딥러닝 네크워크의 크기를 더 늘리는 것입니다. 두번째 아이디어는 더 작은 batch size에서 잘 작동시키기 위해 기존의 group normalization 방법 대신 batch normalization 방법을 사용한 점입니다. 이외에도 attention mechanism등을 네트워크에 추가도 해보았지만 큰 성능향상은 없었습니다. 최종 결과는 5개의 기존 모델과 5개의 개선된 모델에서의 결과를 결합하여 예측하였으며, 간단한 수정사항들만으로도 큰 성능향상을 얻을 수 있었습니다.
My approach to this competition is improving the model from last year winner. It is convenient that the authors provided the codes for their models, significantly simplify the development process. Starting from the baseline model of last year, I added two modifications. The first one is to increase the size of the network because the training data for this year is 4 times more. The second idea is to use group normalization instead of batch normalization since group normalization works better for smaller batch size. I tried other modifications such as incorporating attention mechanism but they did not improve the performance. The final model combined the prediction from 5 baseline and 5 modified models to generate the prediction. The simple modifications were surprisingly effective.
Q4. 이번 challenge를 진행하며 가장 까다로웠던, 혹은 힘들었던 부분이 있으셨는지요? 있으셨다면 어떻게 해결해 나가신지도 궁금합니다.
가장 힘들었던 부분은 개발을 준비하는 단계였던 것 같습니다. 딥러닝 네트워크의 train/validation을 위해 제공된 MRI 데이터 형식을 이해하고 기존 코드들을 분석하는 단계에서 가장 시간과 노력이 많이 투입되었습니다. 또한 3D 네트워크 훈련을 위해 GPU 메모리를 최적화하는 과정, 서로 다른 서버로부터 cross-validation한 결과를 분석하는 과정, 가장 좋은 성능을 보이는 모델을 선정하는 과정도 쉽지 않았던 것 같습니다. 이번 대회의 주어진 시간에 비해 모델을 훈련시키는데에 시간이 많이 걸려서 다양한 수정 사항들을 테스트해보지 못했던 것이 아쉬웠고, 하나의 모델을 주최측에 제출하면 해당 모델의 성능을 주최측 서버에서 평가해주는데 길게는 몇 시간도 걸려 시간이 촉박했던 것 같습니다. 하지만 이번 대회를 통해 뇌종양 데이터를 처리하는 방법과 후처리 방법에 대해 많이 배우게 되었습니다. 또한 이런 대회 진행에 필요한 모델 개발 및 검증 방법, 검증 시간 최적화, 최적의 모델 선택방법 및 이를 정리하여 보고하는 방법등 일반적인 딥러닝 개발 워크 플로우에 익숙해진 것이 앞으로 저에게 큰 자산이 될 것 같습니다.
The beginning of the competition was the hardest part of the process. Setting up the baseline was time-consuming because I needed to understand the data format and the codes for training and evaluating the networks. Other difficulties include fitting the 3D network in GPU memory, aggregating the cross-validation results from multiple different machines, and selecting the best models. The training time of one model was quite long compared to the competition’s timeframe, limiting the space of the modification that can be explored. Evaluation of the model on the validation data was done by the organizers and can be quite slow. I usually had to wait for a few hours for the validation results to move on with the development. I learned a lot about dealing with brain tumor data such as pre or post processing. I also familiarize myself with the typical competition’s workflow such as developing and validating models with cross-validation, test time augmentation and ensembling, and selecting and submitting the optimal model.
Q5. 앞으로 더 연구해보고 싶은 방향은 어떻게 되시나요?
뇌종양 구획 분할과 관련하여 attention-based, 혹은 transformer 모델을 더 연구해보고 싶습니다. 이런 모델들은 기존의 컴퓨터 비전 분야에서 convolutional model에 비해 더 좋은 성능을 보이고 있습니다. 특히 transformer 모델은 의료 데이터에서 좋은 성능을 발휘할 것이라고 생각하지만, 기존의 U-net 기반 모델이 오랜 기간 최적화되었기 때문에 transformer 모델도 모델 구조, 훈련 방법등에서 더 많은 최적화가 필요할 것으로 생각이 되어 이런 방면에서 더 연구를 진행해보고 싶습니다. 또한 딥러닝을 활용하여 MRI 영상을 정량적으로 분석하는 분야도 진행하고 싶습니다. 감사합니다.
With regard to brain tumor segmentation, I want to explore attention-based or transformer models. These models have been shown to perform on-par or better than convolutional models for computer vision tasks. I think transformer models can also provide better performance for medical data, but that would require more optimization of the model architectures or training methods as the current leading models based on UNet has been optimized over several years. For other research, I want to focus on my research interest of quantitative MR imaging with deep learning. Thank you for having me.
참고: Luu, Huan Minh, and Sung-Hong Park. "Extending nn-UNet for brain tumor segmentation." arXiv preprint arXiv:2112.04653 (2021).
김준희 기자 (jjoon95@kaist.ac.kr)