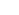
<암세포 유전체 정보 및 분자 네트워크 동역학 분석을 통한 약물 타겟 제시>
1. 안녕하세요, 먼저 자기소개 부탁드립니다.
안녕하세요. 저는 카이스트 바이오및뇌공학과 조광현 교수님 연구실 (시스템생물학 및 바이오영감공학 연구실)의 박사과정 학생 이종훈입니다.
2. 이번에 Biomolecules지에 새로운 연구 결과를 발표하셨는데요. 발표하신 연구에 대해 간단하게 소개해주실 수 있으실까요?
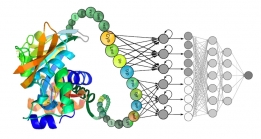
최근 많은 연구가 이루어지고 있는 항암 분자표적치료는 암세포의 특정 유전자 변형에 의해 과활성화 된 신호전달 단백질을 선택적으로 저해하여 암세포의 사멸을 유도하려는 방법입니다. 하지만, 암세포내 분자네트워크의 복잡성과 환자의 유전자 변형의 이질성 때문에 이러한 저해제의 효과는 직관적이지 않으며 환자간 차이도 매우 큽니다. 이번 연구는 이러한 한계를 극복하기 위해 암세포의 유전체 데이터에 기반해 가상 폐암세포 분자조절 네트워크에 대한 수학모형을 만들고 이를 활용한 대규모 컴퓨터 시뮬레이션을 수행하여 암세포주 별 상이한 항암제 반응을 예측할 뿐 아니라 새로운 항암 약물 타겟 후보를 제시하는 분석 방법을 정립하였습니다. 이를 FGFR1 유전자 증폭을 가진 폐암세포주의 FGFR 저해제 반응 예측에 적용하여 약물반응을 결정하는 핵심 분자 메커니즘을 찾고 내성 세포주의 경우 최적의 조합 약물 타겟 후보를 제시하였습니다.
3.컴퓨터 시뮬레이션으로 항암제의 반응을 예측하고 최적의 약물 조합을 찾는다는게 굉장히 흥미로운데요, 이번 연구가 기존의 예측 모델에 비해 갖는 장점은 무엇인가요?
세포는 끊임없이 변하는 환경으로부터의 신호를 입력으로 받아 적절한 출력을 생성함으로써 환경 변화에 반응하는 동적 시스템입니다. 이러한 세포의 반응은 시간에 따라 변화하는 외부 입력과 시스템을 구성하는 요소들 간 복잡하고 비선형적인 상호작용 네트워크에 의해 결정됩니다. 암세포의 유전자변이는 해당 유전자의 기능에 영향을 줄 뿐 아니라 이러한 세포내 신호전달 네트워크에 존재하는 여러 피드백과 크로스톡(crosstalk) 등을 통해 분자네트워크 전반에 걸쳐 고유한 비선형 동역학 특성의 변화를 유발합니다. 이로 인해 다수의 이질적인 유전자 변이를 가지는 암세포들은 각기 고유한 신호전달 동역학 특성을 지니게 되며, 결국 동일 약물에 대해서도 상이한 반응을 보이게 됩니다. 통계분석에 기반한 기존 바이오마커 기반의 항암치료 전략은 이런 복잡한 메커니즘을 반영하기 어렵습니다. 본 연구를 통해, 분자표적 저해제 반응을 체계적으로 분석하기 위해서는 세포를 하나의 시스템으로 바라보고 구성요소들의 상호관계와 상호작용을 분석하는 시스템 생물학(Systems biology) 관점의 접근이 필요함을 강조하고 싶습니다. 본 연구에서 제시하는 동역학 분석 파이프라인은 폐암 뿐 아니라 대장암, 유방암 등 주요 암과 노화 및 뇌 질환 등 여러 질병모델에 적용하여 분자 메커니즘 기반의 새로운 약물 타겟 후보를 탐색하는 도구로 사용될 수 있을 것이라 생각합니다.
4.자세한 설명 감사합니다. 혹시 개인적으로 더 연구하고 싶은 주제나 분야가 있으신지요?
최근 암 관련 수많은 단일세포 다중 오믹스 데이터가 생산되고 있습니다. 하지만, 이러한 데이터를 분자 네트워크 동역학에 기반해 체계적으로 분석할 수 있는 방법은 아직 부족한 실정입니다. 생물학적 사전지식들과 다양한 단일세포 오믹스 데이터를 통합하여 시뮬레이션이 가능한 동역학 모델을 생성하고 이를 기반으로 세포의 상태를 제어하는 타겟 발굴을 위한 연구를 진행해보면 좋을 것 같습니다.
지금까지 카이스트 바이오및뇌공학과 조광현 교수님 연구실 박사과정 학생 이종훈이었습니다. 인터뷰에 응해주셔서 감사드립니다!
-
 [학과 교수 취재] 박은영 교수
[학과 교수 취재] 박은영 교수<박은영 교수> 이번 달에는 올해 우리 학과에 새로 부임하신 박은영 교수님을 취재하였습니다. 박은영...
2024-03-29 -
 [학과 연구성과 취재] 최예진 석사과정 학생
[학과 연구성과 취재] 최예진 석사과정 학생<최예진 석사과정 학생> 이번 달에는 2023 한국바이오칩학회 추계학술대회에서 우수 포스터 발표상을 ...
2024-02-28 -
 [과학 산책] 최정균 교수님, 오믹스 데이터를 기반으로...
[과학 산책] 최정균 교수님, 오믹스 데이터를 기반으로...Figure 1 최정균 교수님 ‘과학 산책’ 시리즈는 교수님과의 인터뷰를 통해 바이오및뇌공학과의 다양한 연구 ...
2024-01-19 -
 [학과 연구성과 취재] 황선하 박사과정 학생
[학과 연구성과 취재] 황선하 박사과정 학생<황선하 박사과정 학생> 이번 달에는 올해 10월 Nature journal의 Scientific Reports에 “Distortion ...
2024-01-02 -
 [학과 교수 취재] 박성준 교수
[학과 교수 취재] 박성준 교수<박성준 교수> 이번 달에는 2023년 우수과학자포상의 젊은 과학자상을 수상하신 박성준 교수님과 인터...
2023-11-27 -
 [학생 인터뷰] 안진현 박사, 딥러닝 모델을 통해 면역 ...
[학생 인터뷰] 안진현 박사, 딥러닝 모델을 통해 면역 ...<연구 관련 내용 대표 이미지> <안진현 박사> 이번 달은 카이스트 바이오및뇌공학과 최정균 교...
2023-10-24 -
 새내기를 위한 학과 선택 가이드
새내기를 위한 학과 선택 가이드https://www.youtube.com/watch?v=_mNbw3_o5jM [새내기를 위한 학과 선택 가이드] 취업 잘 되는 과로 가는게...
2023-09-07 -
 [학과 교수 취재] 이혜선 교수
[학과 교수 취재] 이혜선 교수<이혜선 교수> 이번 달에는 올해 6월 우리 학과에 새로 부임하신 이혜선 교수님을 취재하였습니다. 이...
2023-09-04 -
 [학과 교수 취재] 강형률 교수
[학과 교수 취재] 강형률 교수<강형률 교수> 이번 인터뷰는 새로이 바이오 및 뇌공학과에 부임하신 강형률 교수님과 진행해보았습니...
2023-09-01 -
 [과학 산책] 정기훈 교수님, ‘BioMEMS/NEMS’ 기술을 기...
[과학 산책] 정기훈 교수님, ‘BioMEMS/NEMS’ 기술을 기...<정기훈 교수님> ‘과학 산책’ 시리즈는 교수님과의 인터뷰를 통해 바이오및뇌공학과의 다양한 연구 분...
2023-08-04 -
 [학과 연구성과 취재] 김정연 박사
[학과 연구성과 취재] 김정연 박사<김정연 박사> 이번 달은 바이오 및 뇌공학과 교수님 연구실에서 연구하고 계신 김정연 박사님과 인터...
2023-08-01 -
 [학과 연구성과 취재] 김효민 박사과정 학생
[학과 연구성과 취재] 김효민 박사과정 학생<김효민 박사과정 학생> 이번 달에는 올해 2월 Aging Cell에 ‘Glycation-mediated Tissue-level Remod...
2023-07-03 -
 [학과 연구성과 취재] 성창훈 박사과정 학생
[학과 연구성과 취재] 성창훈 박사과정 학생<성창훈 박사과정 학생> 이번 달에는 올해 4월 Nature Communications에 ‘Highly conductive tissue-l...
2023-07-03 -
 [학과 교수 취재] 신우정 교수
[학과 교수 취재] 신우정 교수<바이오및뇌공학과 신우정 교수> 이번 달에는 올해 3월 우리 학과에 새로 부임하신 신우정 교수님을 ...
2023-05-31 -
 [학과 연구 성과] 권준하 선생님
[학과 연구 성과] 권준하 선생님<권준하 박사님> 이번 달은 바이오 및 뇌공학과 최정균 교수님 연구실에서 연구하고 계신 권준하 박사...
2023-05-29 -
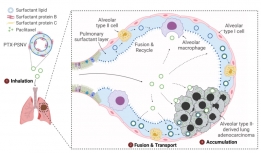 [학생 인터뷰] 오찬희 박사과정생, 나노입자를 이용한 ...
[학생 인터뷰] 오찬희 박사과정생, 나노입자를 이용한 ...<그림. 오찬희 박사과정분이 개발한 새로운 흡입성 폐암치료제의 작용기작> 오늘은 바이오 및 뇌공학...
2023-04-27 -
 [학과 교수 취재] 정아인 교수
[학과 교수 취재] 정아인 교수<정아인 교수> 이번 인터뷰는 새로이 바이오 및 뇌공학과에 부임하신 정아인 교수님과 진행해보았습니...
2023-04-27 -
 [학과 교수 취재] 손성민 교수
[학과 교수 취재] 손성민 교수<손성민 교수> 이번 달에는 작년 7월 우리 학과에 새로 부임하신 손성민 교수님을 취재하였습니다. 손...
2023-03-30 -
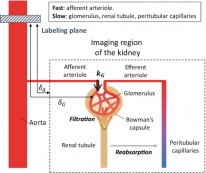 [학생 인터뷰] 확산 강조 동맥스핀표지 기법을 사용한 ...
[학생 인터뷰] 확산 강조 동맥스핀표지 기법을 사용한 ...<사구체 혈액 전달 속도 분석 모델> 1. 안녕하세요, 먼저 자기소개 부탁드립니다. 안녕하세요. 바이오...
2023-02-23 -
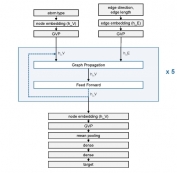 [학생 인터뷰] 김하영 박사과정생, 단백질 복합체 구조...
[학생 인터뷰] 김하영 박사과정생, 단백질 복합체 구조...<스코어링 모델> 1. 안녕하세요, 자기소개 부탁드립니다. 안녕하세요, 바이오및뇌공학과 김동섭 교수...
2023-02-22 -
 [과학산책] 박성홍 교수, 뇌 노폐물 배출의 영상화
[과학산책] 박성홍 교수, 뇌 노폐물 배출의 영상화바이오및뇌공학과의 다양한 연구 분야를 교수님과의 인터뷰를 통해 알아보는 ‘과학 산책’ 시리즈입니다. 이...
2023-02-01 -
 [학생 인터뷰] 김정연 박사과정생, mRNA 암백신 핵심기...
[학생 인터뷰] 김정연 박사과정생, mRNA 암백신 핵심기...<mRNA 암백신 핵심기술> Q1. 안녕하세요, 자기소개 부탁드립니다. 안녕하세요. 저는 카이스트 바이오...
2023-01-19 -
 [학생 인터뷰] 2022년 가을학기 학과 상담 조교, 윤주...
[학생 인터뷰] 2022년 가을학기 학과 상담 조교, 윤주...<바이오및뇌공학과> 1. 인터뷰에 응해주셔서 감사합니다. 이번 인터뷰에서는 학과 학생들의 여러가지 ...
2023-01-02 -
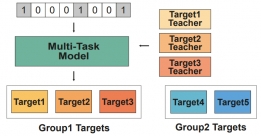 [학생 인터뷰] 문채영 박사과정생, 다중작업학습을 통...
[학생 인터뷰] 문채영 박사과정생, 다중작업학습을 통...<다중작업학습을 통한 저분자 화합물과 단백질 결합 예측> Q1. 자기소개 부탁드립니다. 안녕하세요, ...
2022-12-28 -
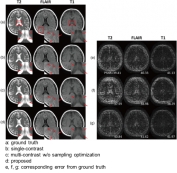 [학생 인터뷰] Huan Minh Luu 박사과정, 다중 대조도 M...
[학생 인터뷰] Huan Minh Luu 박사과정, 다중 대조도 M...<모델 기반 딥러닝 활용 샘플링 패턴 최적화> Q1. 인터뷰에 응해주셔서 감사드립니다! 먼저 자기소개 ...
2022-12-01 -
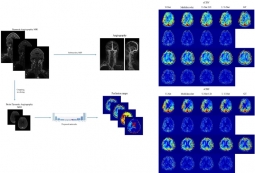 [학생 인터뷰] Muhammad 박사과정생, 혈관 MRI 이미지...
[학생 인터뷰] Muhammad 박사과정생, 혈관 MRI 이미지...<혈관 MRI 이미지를 이용한 관류 MRI 이미지 예측 모델 > Q1. 인터뷰에 응해주셔서 감사드립니다! 먼...
2022-11-29 -
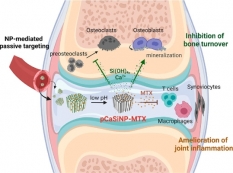 [학생 인터뷰] 정문경 박사 후 과정 연구원, 류마티스 ...
[학생 인터뷰] 정문경 박사 후 과정 연구원, 류마티스 ...<류마티스 관절염 차료용 다공성 실리콘 입자 기반 약문 전달 시스템> Q1. 안녕하세요, 먼저 자기소개...
2022-10-31 -
 [학생 인터뷰] 권준하 박사, 유전체 분석을 통한 공격...
[학생 인터뷰] 권준하 박사, 유전체 분석을 통한 공격...<공격성 섬유종증의 화학적 항암요법 치료 효과 예측 모델> Q1. 인터뷰에 응해주셔서 감사드립니다! ...
2022-10-31 -
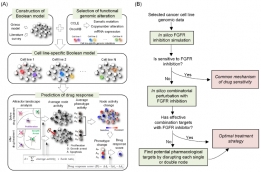 [학생인터뷰] 이종훈 박사과정 학생, 암세포 유전체 정...
[학생인터뷰] 이종훈 박사과정 학생, 암세포 유전체 정...<암세포 유전체 정보 및 분자 네트워크 동역학 분석을 통한 약물 타겟 제시> 1. 안녕하세요, 먼저 자...
2022-10-04 -
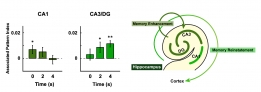 [학생 인터뷰] 강준영 박사, 기억인출 과정동안 뇌의 ...
[학생 인터뷰] 강준영 박사, 기억인출 과정동안 뇌의 ...<뇌의 해마에서 나타나는 기억 강화 관련 신경 표상> Q1. 인터뷰에 응해주셔서 감사드립니다! 먼저 자...
2022-09-26 -
 [학생 인터뷰] 이정민 박사, ‘가십’ 행위가 사회적 목...
[학생 인터뷰] 이정민 박사, ‘가십’ 행위가 사회적 목...<사회적 정보를 인풋으로 받아서 여러 내부 프로세스를 거쳐 처리되는 과정을 담은 이론 모델> 이번 ...
2022-08-30 -
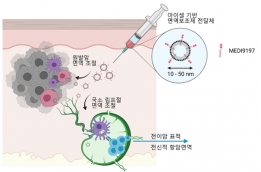 [학생 인터뷰] 정문경 박사과정 학생, 지질나노입자를 ...
[학생 인터뷰] 정문경 박사과정 학생, 지질나노입자를 ...<연구 내용 관련 대표 이미지> Q1. 인터뷰에 응해주셔서 감사드립니다! 먼저 자기소개 부탁드립니다. ...
2022-08-26 -
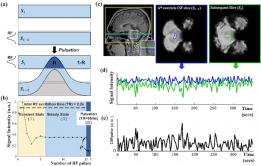 [학생 인터뷰] 임재근 박사과정생, 뇌 활동 중 뇌척수...
[학생 인터뷰] 임재근 박사과정생, 뇌 활동 중 뇌척수...<연구 내용 관련 대표 이미지> 이번 달에는 최근 “Measurement of CSF pulsation from EPI-based huma...
2022-08-01 -
 [과학산책] 김동섭 교수, 구조생물학과 AI
[과학산책] 김동섭 교수, 구조생물학과 AI<구조생물학과 AI〉 바이오및뇌공학과의 다양한 연구 분야를 교수님과의 인터뷰를 통해 알아보는 ‘과학 ...
2022-08-01 -
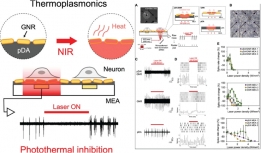 [학생 인터뷰] 윤동조 박사과정생, 폴리도파민 화학적 ...
[학생 인터뷰] 윤동조 박사과정생, 폴리도파민 화학적 ...<연구 내용 관련 대표 이미지> 이번 달에는 최근 “Enhancement of Thermoplasmonic Neural Modulati...
2022-06-29 -
 [학생 인터뷰] 방효은 박사과정 학생, 항암 면역반응을...
[학생 인터뷰] 방효은 박사과정 학생, 항암 면역반응을...<연구 내용 관련 대표 이미지> Q1. 인터뷰에 응해주셔서 감사드립니다! 먼저 자기소개 부탁드립니다. ...
2022-06-24 -
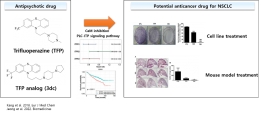 [학생 인터뷰] 박항익 박사과정생, 향정신병제제인 tri...
[학생 인터뷰] 박항익 박사과정생, 향정신병제제인 tri...<연구 내용 관련 대표 이미지> 이번 달에는 최근 “Trifluoperazine and Its Analog Suppressed the Tu...
2022-05-30 -
 [학생 인터뷰] 강의룡 박사과정 학생, 브레인 네트워크...
[학생 인터뷰] 강의룡 박사과정 학생, 브레인 네트워크...<연구 내용 관련 대표 이미지> 1. 안녕하세요, 먼저 자기소개 부탁드립니다. 안녕하세요. 저는 카이스...
2022-05-25 -
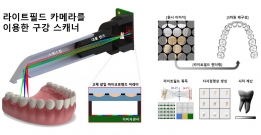 [학생 인터뷰] 권재명 석사과정 학생, 3D 생체 이미징 ...
[학생 인터뷰] 권재명 석사과정 학생, 3D 생체 이미징 ...<연구 관련 내용 대표 이미지> 이번 학생 인터뷰에서는 3D 생체 이미징을 개발하고 이를 구강 스캐너...
2022-05-02 -
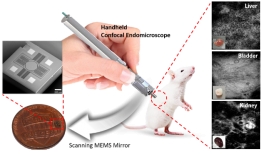 [학생 인터뷰] 전재훈 석사과정생, 고속 이미징이 가능...
[학생 인터뷰] 전재훈 석사과정생, 고속 이미징이 가능...<연구 내용 관련 대표 이미지> 이번 달에는 최근 “Handheld laser scanning microscope catheter for ...
2022-05-02 -
 [학생 인터뷰] 박성용 박사과정 학생, 삼중음성 유방암...
[학생 인터뷰] 박성용 박사과정 학생, 삼중음성 유방암...<연구 내용 관련 대표 이미지> 이번 학생 인터뷰에서는 최근에 국제 학술지 ‘Cancers’에 게재된 삼중...
2022-03-29 -
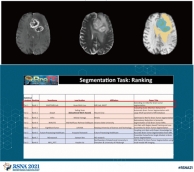 [학생 인터뷰] Luu Minh-Huan 박사과정생, MICCAI/ASNR...
[학생 인터뷰] Luu Minh-Huan 박사과정생, MICCAI/ASNR...<본 연구팀이 제안한 뇌종양 구획화 모델 대표 이미지(위), BraTS 대회 딥러닝 모델성능 순위(아래)> ...
2022-03-28 -
 [학생인터뷰] 이도헌 교수님 연구팀, Heart Disease AI...
[학생인터뷰] 이도헌 교수님 연구팀, Heart Disease AI...<임수린, 김관수, 김유태, 한만영 학생의 Heart Disease AI Datathon 2021 대회 수상 장면> 바이오및...
2022-03-03 -
 [학생 인터뷰] 김동재 박사 졸업생, 인공지능의 오랜 ...
[학생 인터뷰] 김동재 박사 졸업생, 인공지능의 오랜 ...<김동재 박사> Q1) 안녕하세요 김동재 박사님, 바쁘신 와중에 인터뷰를 허락해 주셔서 감사드립니다. ...
2022-02-07 -
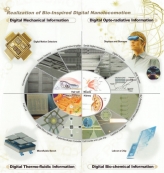 [과학산책] 생체모사 디지털나노시스템에 대하여
[과학산책] 생체모사 디지털나노시스템에 대하여〈생체모사 디지털나노시스템〉 바이오및뇌공학과의 다양한 연구 분야에 대해 교수님과의 인터뷰를 통해 알...
2022-01-21 -
 [학생 인터뷰] 김수지 박사과정 학생, 새로운 바이오 ...
[학생 인터뷰] 김수지 박사과정 학생, 새로운 바이오 ...〈연구 내용 관련 대표 이미지〉 이번 학생 인터뷰에서는 최근 국제 학술지 ‘Advanced Materials Technologi...
2022-01-13 -
 [학생 인터뷰] 최새롬 박사과정 학생, 암세포의 성질을...
[학생 인터뷰] 최새롬 박사과정 학생, 암세포의 성질을...〈최새롬 박사과정〉 Q1) 안녕하세요 최새롬 선생님, 바쁘신 와중에 인터뷰를 허락해 주셔서 감사드립니다. ...
2022-01-05 -
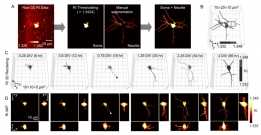 [학생 인터뷰] 윤동조 박사과정 학생, 비표지 3차원 신...
[학생 인터뷰] 윤동조 박사과정 학생, 비표지 3차원 신...〈3차원으로 촬영된 신경세포의 발달과정〉 이번 학생 인터뷰에서는 최근 국제 학술지 ‘Biomedical Optics E...
2021-12-02 -
 [학생 인터뷰] 장재선 박사 졸업생, 다학제적 접근 통...
[학생 인터뷰] 장재선 박사 졸업생, 다학제적 접근 통...<장재선 박사 졸업생> Q1) 안녕하세요 장재선 박사님, 바쁘신 와중에 인터뷰를 허락해 주셔서 감사드...
2021-12-02 -
 [학생 인터뷰] 배상인 박사 졸업생, 4D 촬영이 가능한 ...
[학생 인터뷰] 배상인 박사 졸업생, 4D 촬영이 가능한 ...<4D 초박형 라이트필드 카메라> 이번 학생 인터뷰에서는 최근 언론과 국제 학술지 ‘Advanced Optical ...
2021-11-02 -
 [학생 인터뷰] 2021년 가을학기 학과 상담 조교장, 배...
[학생 인터뷰] 2021년 가을학기 학과 상담 조교장, 배...<바이오및뇌공학과> 이번 달에는 이번 학기 저희 학과 상담 조교장을 맡고 계신 배효경 님과 인터뷰를...
2021-10-27 -
 [학생 인터뷰] 석민호 박사과정 학생, 숨 쉬는 웨어러...
[학생 인터뷰] 석민호 박사과정 학생, 숨 쉬는 웨어러...다공성 헤어셀 구조의 맥파센서 : (a) 맥파센서 상단면 현미경 사진; (b) 헤어셀 높이별(0um, 50um, 100um) ...
2021-09-27 -
 [학과 교수 취재] 박성준 교수, 하이드로젤 기반 유연...
[학과 교수 취재] 박성준 교수, 하이드로젤 기반 유연...하이드로젤 기반 유연성 뇌-기계 인터페이스 Q1) 안녕하세요 박성준 교수님, 바쁘신 와중에 인터뷰를 허락해...
2021-09-15 -
 [학생 인터뷰] 장경원 박사과정 학생, 곤충눈 모사 초...
[학생 인터뷰] 장경원 박사과정 학생, 곤충눈 모사 초...<장경원 마이크로픽스 대표> 이번 달에는 곤충눈 모사 초박형 카메라에 관해 연구하시고 해당 기술로 ...
2021-09-06 -
 [학생 인터뷰] 송준영 박사과정 학생, 인공위성 영상의...
[학생 인터뷰] 송준영 박사과정 학생, 인공위성 영상의...<송준영 박사과정 학생> 이번달에는 IEEE Trans. on Geoscience and Remote Sensing에 발표된 송준영 ...
2021-08-31 -
 [학과 교수 취재] 백세범 교수, 카이스트 싱귤레러티 ...
[학과 교수 취재] 백세범 교수, 카이스트 싱귤레러티 ...<백세범 교수> 이번 달에는 카이스트에서 처음 시도하는 임용 후 10~20년간 논문 평가를 받지 않는 싱...
2021-08-31 -
 [과학산책] 조광현 교수님, 역노화 원천기술 개발
[과학산책] 조광현 교수님, 역노화 원천기술 개발<조광현 교수> 이번 [과학산책]에서는 바이오및뇌공학과 조광현 교수님과 인터뷰를 진행하였습니다. ...
2021-07-12 -
 [학과 교수 취재] 이상완 교수, IBM 학술상 수상
[학과 교수 취재] 이상완 교수, IBM 학술상 수상<이상완 교수> 이번 달에는 미국 IBM과 전 세계 유수 대학과의 연구 협력 활성화를 위해 제정된 상인 ...
2021-07-12 -
 [학생 인터뷰] 박영진 박사과정 학생, 고효율 물체인식...
[학생 인터뷰] 박영진 박사과정 학생, 고효율 물체인식...<박영진 박사과정 학생> 이번달에는 Neural Networks에 발표된 박영진 박사과정 학생의 물체 인식에 ...
2021-07-05 -
 [봄학기 CA 취재] 2021년 봄학기 학과 상담 조교장, 장...
[봄학기 CA 취재] 2021년 봄학기 학과 상담 조교장, 장...<바이오및뇌공학과> 이번달에는 이번학기 저희 학과 상담 조교장을 맡고 계신 장민철 님과 인터뷰를 ...
2021-06-18 -
 [학생 인터뷰] 강병훈 박사과정 학생, 실시간 나노플라...
[학생 인터뷰] 강병훈 박사과정 학생, 실시간 나노플라...(좌) 강병훈 박사과정 학생 (우) 정기훈 교수 실시간 나노플라즈모닉 PCR을 통한 초고속 분자진단 시스템 이...
2021-06-10 -
 [학과 교수 취재] 이수현 교수, 뇌 복부선조영역의 새...
[학과 교수 취재] 이수현 교수, 뇌 복부선조영역의 새...<이수현 교수> 이번 달에는 이수현 교수님과 4월 8일 Nature Communications에 개재된 연구 (논문명: ...
2021-05-06 -
 [학과 교수 취재] 이영석 교수
[학과 교수 취재] 이영석 교수<이영석 교수> 이번달에는 바이오및뇌공학과에 새로 부임하신 이영석 교수님을 취재하였습니다. 이영...
2021-04-14 -
 [학과 교수 취재] 박성준 교수, 인공근육 재생을 위한 ...
[학과 교수 취재] 박성준 교수, 인공근육 재생을 위한 ...이번 달에는 박성준 교수님과 2월 19 Advanced Material에 출판된 연구 (논문명: Functional skeletal muscl...
2021-04-01 -
 [졸업생 취재] 이영범박사 (IBS 인지 및 사회성연구단)
[졸업생 취재] 이영범박사 (IBS 인지 및 사회성연구단)<이영범 박사님> KAIST 바이오및뇌공학과에서 박사 과정을 마치시고 현재 기초과학연구원(IBS)에서 근...
2021-02-26 -
 [학과 교수 취재] 최철희 교수, 엑소좀(Exosome)기반 ...
[학과 교수 취재] 최철희 교수, 엑소좀(Exosome)기반 ...<최철희 교수> 이번 연구 성과 취재에서는 바이오및뇌공학과 교수이자 ‘일리아스바이오로직스’의 CEO...
2021-02-26 -
 [학생 인터뷰] 장용희, 조재욱 예비 창업팀, 쥐 뇌파 ...
[학생 인터뷰] 장용희, 조재욱 예비 창업팀, 쥐 뇌파 ...< (왼쪽부터) 장용희, 조재욱 석사과정 > 이번에는 카이스트 바이오및뇌공학과/뇌인지공학 프로그램 ...
2021-01-29 -
 [학생 인터뷰] 허재영, Shujaat Khan 예비창업팀, 딥러...
[학생 인터뷰] 허재영, Shujaat Khan 예비창업팀, 딥러...< (왼쪽부터) 허재영, Shujaat Khan 박사과정 > 이번에는 카이스트 바이오및뇌공학과/뇌인지공학 프로...
2021-01-04 -
 [학생 인터뷰] 이동구, 송국호, 이찬석 예비창업팀, 휴...
[학생 인터뷰] 이동구, 송국호, 이찬석 예비창업팀, 휴...<(왼쪽부터) 이동구 박사과정, 이찬석 석사과정, 송국호 석사과정 > 이번에는 카이스트 바이오및뇌공...
2021-01-04 -
 [학생 인터뷰] 이남주, 김한진, 박지성 예비창업팀, ‘...
[학생 인터뷰] 이남주, 김한진, 박지성 예비창업팀, ‘...<(왼쪽부터) 이남주 박사과정, 김한진 석사과정, 박지성 석사과정> 이번에는 카이스트 바이오및뇌공학...
2021-01-04 -
 [학생 인터뷰] 이준철 박사 과정생, 튜불린을 이용한 ...
[학생 인터뷰] 이준철 박사 과정생, 튜불린을 이용한 ...<(왼쪽부터)이준철 박사과정, Advanced Materials 표지 이미지, Small 후면 표지 이미지> 이번 연구 ...
2020-12-01 -
 [과학산책] 이상완 교수, 딥러닝(Deep Learning)으로 ...
[과학산책] 이상완 교수, 딥러닝(Deep Learning)으로 ...<이상완 교수> 이번 [과학산책] 인터뷰는 바이오 및 뇌공학과 이상완 교수님과 진행하였습니다. Q1. ...
2020-12-01 -
 [학과 교수 취재] 박영균 교수
[학과 교수 취재] 박영균 교수<박영균 교수> “누구도 가지 않았던 길을 가십시오.” 이번에 새로이 바이오및뇌공학과에 부임하신 박...
2020-11-02 -
 [학생 인터뷰] 오유진 박사 과정생
[학생 인터뷰] 오유진 박사 과정생<오유진 박사과정생 (가운데)> Q1. 우선 간단한 자기소개 부탁드립니다. 안녕하세요, 저는 예종철 교...
2020-09-21 -
 [학생 인터뷰] 김희곤 박사 후 과정 연구원
[학생 인터뷰] 김희곤 박사 후 과정 연구원<김희곤 박사 후 과정 연구원> Q1. 우선 간단한 자기소개 부탁드립니다. 안녕하세요, 저는 바이오및뇌...
2020-09-21 -
 [학생 인터뷰] 2020년 봄학기 학과 상담 조교장. 김가...
[학생 인터뷰] 2020년 봄학기 학과 상담 조교장. 김가...Q1. 간단한 자기소개 부탁 드립니다. 안녕하세요. 저는 바이오 및 뇌공학과에서 학사, 석사를 졸업하고 현재...
2020-08-31 -
 [학생 인터뷰] 김진우 학부생, 시각 피질의 주요 신경...
[학생 인터뷰] 김진우 학부생, 시각 피질의 주요 신경...Q1. 간단한 자기소개 부탁드립니다. 안녕하세요, 바이오 및 뇌공학과와 전산학부를 복수 전공했고 이제 졸업...
2020-08-31 -
 [학생 인터뷰] 오찬희 박사과정생, 혈관 조영제로 폐암...
[학생 인터뷰] 오찬희 박사과정생, 혈관 조영제로 폐암...이번 달에는 혈관 조영제로 폐암 종양 절제부위를 정확하게 구분하는 기술을 개발하신 박지호 교수님 연구실...
2020-08-03 -
 [학생 인터뷰] 최우철 박사과정생, 뇌 절편 영상 자동 ...
[학생 인터뷰] 최우철 박사과정생, 뇌 절편 영상 자동 ...이번 달에는 최근 실험용 쥐의 뇌 절편 영상을 자동으로 보정하고 규격화하여 신경세포의 3차원 분포정보를 ...
2020-07-10 -
 [학과 교수 취재] 백세범 교수
[학과 교수 취재] 백세범 교수이번 달에는 백세범 교수님과의 인터뷰를 진행해보았습니다. 백 교수님은 캘리포니아 대학교 버클리 (UC Ber...
2020-06-01 -
 [학생 인터뷰] 김정연 석사과정생, 면역항암치료의 bio...
[학생 인터뷰] 김정연 석사과정생, 면역항암치료의 bio...이번 달에는 최근 Nature Communications에 논문을 발표하신 최정균 교수님 연구실의 김정연 석사과정 학생...
2020-05-28 -
 [학과 교수 취재] 박지호 교수
[학과 교수 취재] 박지호 교수이번 달에는 생체 재료 분야에서 나노 의학 연구를 주로 진행하고 계시는 박지호 교수님과 인터뷰를 진행하...
2020-05-04 -
 [학생 인터뷰] 서성훈 박사과정생, 딥러닝 통해 MRI 다...
[학생 인터뷰] 서성훈 박사과정생, 딥러닝 통해 MRI 다...(왼쪽부터 1저자 도원준 박사, 공동 1저자 서성훈 박사과정, 지도교수 박성홍 교수님) 이번 달에는 최근 Med...
2020-04-28 -
 [학생 인터뷰] 장재선, 송민 박사과정생, 포유류 시각...
[학생 인터뷰] 장재선, 송민 박사과정생, 포유류 시각...이번 달에는 최근 Cell Reports에 논문을 발표하신 백세범 교수님 연구실의 장재선, 송민 박사과정 학생분들...
2020-04-01 -
 [학과 교수 취재] 김동섭 교수
[학과 교수 취재] 김동섭 교수이번 달에는 생물정보학(Bioinformatics) 분야에서 단백질의 구조와 서열에 대한 연구를 주로 진행하고 계시...
2020-03-11 -
 [학과 교수 취재] 예종철 교수
[학과 교수 취재] 예종철 교수이번달에는 IEEE Fellow 선정, Nature Machine Intelligence 게재, IEEE EMBS Distinguished Lecturer 선정...
2020-02-27 -
 [학생 인터뷰] 김동재 박사과정생, 메타 강화학습 뇌 ...
[학생 인터뷰] 김동재 박사과정생, 메타 강화학습 뇌 ...이번 달에는 뇌인지공학프로그램 박사과정 김동재 학생 취재하였습니다. 김동재 박사과정생은 Caltech과 메...
2020-02-06 -
 [과학산책] 정용 교수
[과학산책] 정용 교수이번 과학산책으로는 뇌/신경분야의 연구를 진행하고 계시는 정 용 교수님과 인터뷰를 진행하였습니다. 1. ...
2020-02-03 -
 [과학산책] 박성홍 교수
[과학산책] 박성홍 교수이번달에는 Biomedical Imaging 분야에서 MRI관련 연구를 진행하고 계시는 박성홍 교수님과 면담을 진행하였...
2020-02-03 -
 [학과 교수 취재] 조광현 교수
[학과 교수 취재] 조광현 교수[조광현 교수] 이번 달에는 시스템 생물학을 연구하시는 조광현 교수님을 취재하였습니다. 조광현 교수님이 ...
2020-01-21 -
 [학생인터뷰] 황경민 현 “VPIX”대표이사
[학생인터뷰] 황경민 현 “VPIX”대표이사이번 인터뷰에서는 바이오 뇌공학과 정기훈 교수님 연구실 박사과정 황경민 현 “VPIX”대표이사님과의 인터뷰...
2019-12-26 -
 [학과 교수 취재] 장무석 교수
[학과 교수 취재] 장무석 교수이번 달에는 장무석 교수님을 취재하였습니다. 장무석 교수님은 카이스트 물리학과 학부를 졸업하시고 2016...
2019-11-13 -
 [과학산책] 박제균 교수
[과학산책] 박제균 교수MicroTAS(micro Total Analysis Systems)는 microelectromechanical systems (MEMS) 또는 microsystems tech...
2019-10-31 -
 [과학산책] 이상완 교수
[과학산책] 이상완 교수[과학산책] 이상완 교수 이번 달에는 KAIST 핵심 기술로 선정된 신경과학-인공지능 융합형 차세대 초고성능 ...
2019-10-20 -
 [학과 교수 취재] 최정균 교수, 새로운 항암면역치료 ...
[학과 교수 취재] 최정균 교수, 새로운 항암면역치료 ...눈문링크: https://www.nature.com/articles/s41467-019-12159-9 이번 달에는 지난 9월 nature communicatio...
2019-10-15 -
 [학생 인터뷰] 김준희 석사과정생, 공동저자로 Nature...
[학생 인터뷰] 김준희 석사과정생, 공동저자로 Nature...이번 달에는 nature에 ‘퇴행성 뇌 질환 유발 물질의 배출경로 규명’의 주제를 가지고 published 된 논문의 ...
2019-09-24 -
 [과학산책] 이수현 교수
[과학산책] 이수현 교수이수현교수님께서는 카이스트 물리학과를 졸업하시고, 서울대학교에서 생명과학 박사학위를 받으시고, 미국 ...
2019-08-20 -
 [학생 인터뷰] 이현수 조교
[학생 인터뷰] 이현수 조교이번 학과 취재 인터뷰는 2019년 봄학기 동안 CA(학사과정 학업 및 진로 상담) 활동하셨던 이현수 CA장을 인...
2019-08-05 -
 [과학산책] 김필남 교수
[과학산책] 김필남 교수Q1. 김필남교수님께서는 최근에 한국 로레알-유네스코 여성과학자상 펠로십을 수상하셨습니다. 펠로십은 앞...
2019-07-25 -
 [졸업생취재] 신중호 교수(현, 부경대학교 의공학과)
[졸업생취재] 신중호 교수(현, 부경대학교 의공학과)신중호 교수님은 University of California, San Diego Dept. of Bioengineering에서 학사 학위를 받으시고 ...
2019-07-11 -
 [학생 인터뷰] Maryam Yavartanoo / Yi Li
[학생 인터뷰] Maryam Yavartanoo / Yi Li이번 달에는 바이오및뇌공학과 대학원 박사과정을 밟고 있는 Maryam Yavartanoo와 Yi Li 학생과의 인터뷰를 ...
2019-06-26 -
 [과학산책] 바이오인포매틱스 (Bioinformatics)
[과학산책] 바이오인포매틱스 (Bioinformatics)'과학산책' 시리즈는 바이오공학과 뇌공학의 다양한 분야에 대하여 카이스트 바이오및뇌공학과 교수님들과 ...
2019-06-07 -
 [졸업생취재] 이옥균 교수 (DGIST 로봇공학전공)
[졸업생취재] 이옥균 교수 (DGIST 로봇공학전공)이옥균 교수님은 KAIST 바이오및뇌공학과 예종철 교수님 연구실에서 석사, 박사 과정을 마치시고 Johns Hopk...
2019-05-27 -
 [학과 교수 취재] 김철 교수
[학과 교수 취재] 김철 교수인터뷰는 4월 22일 교수님의 오피스에서 진행되었다. 열려있는 문으로 가볍게 노크를 하고 들어가자 교수님...
2019-04-25 -
 [학과 교수 취재] 박성준 교수
[학과 교수 취재] 박성준 교수박성준 교수님은 서울대학교 기계항공공학부에서 학사, MIT기계공학과에서 석사, 동 대학 전기컴퓨터공학과...
2019-04-15 -
 [학과 교수 취재] 정재승 교수
[학과 교수 취재] 정재승 교수안녕하세요. 교수님 일단 이렇게 서면 인터뷰에 응해주셔서 매우 감사합니다. 아래에 준비된 질문 문항들이 ...
2019-04-10 -
 [졸업생취재] 김상우 교수 (현, 연세대학교 의과대학 ...
[졸업생취재] 김상우 교수 (현, 연세대학교 의과대학 ...김상우 교수님은 전자전산학과에서 학사, 바이오및뇌공학과(전, 바이오시스템학과)에서 석사, 박사학위를 받...
2019-01-29 -
 [졸업생취재] 나도균 교수 (현, 중앙대학교)
[졸업생취재] 나도균 교수 (현, 중앙대학교)인터뷰에 앞서 나도균 교수님의 약력은 다음과 같습니다. • Mar 1996 – Feb 2000 B.S. Korea University, Se...
2019-01-28 -
 [졸업생취재] 정동일 교수 (현, UNIST 인간공학과)
[졸업생취재] 정동일 교수 (현, UNIST 인간공학과)정동일 교수님은 바이오및뇌공학과(전, 바이오시스템학과)에서 학사, 박사학위를 받으시고, 6년간 버지니아...
2019-01-03 -
 [졸업생취재] 최성용교수(현, 경희대학교 생체의공학과)
[졸업생취재] 최성용교수(현, 경희대학교 생체의공학과)인터뷰 기사에 앞서 최성용 교수님의 약력은 다음과 같습니다. 1999. 3. ~ 2003. 8 B.S. Department of Me...
2018-12-20 -
 [졸업생취재] 김민석 교수 (현, DIGST 뉴바이올로지학과)
[졸업생취재] 김민석 교수 (현, DIGST 뉴바이올로지학과)김민석 교수님은 바이오및뇌공학과(전, 바이오시스템학과)에서 석사, 박사학위를 받으시고, 6년간 삼성종합...
2018-12-05 -
 [졸업생취재] 강주헌 교수(현, UNIST Biomedical Engin...
[졸업생취재] 강주헌 교수(현, UNIST Biomedical Engin...인터뷰 기사에 앞서 강주헌교수님의 약력은 다음과 같습니다. · Assistant Professor, Department of Biomed...
2018-11-26 -
 [졸업생취재] 남호정 교수 (현, GIST 전기전자컴퓨터공...
[졸업생취재] 남호정 교수 (현, GIST 전기전자컴퓨터공...남호정 교수님은 전산학과에서 석사 학위, 바이오및뇌공학과(PI 이도헌 교수님)에서 박사 학위를 받으시고 4...
2018-11-01 -
 [졸업생취재] 최명환 교수(현, 성균관대 글로벌바이오...
[졸업생취재] 최명환 교수(현, 성균관대 글로벌바이오...인터뷰 기사에 앞서 최명환교수님의 약력은 다음과 같습니다. 03/2015~present Assistant professor Departm...
2018-10-26 -
 [학과 교수 취재] 조영호 교수, 『이것이 이공계다』 출간
[학과 교수 취재] 조영호 교수, 『이것이 이공계다』 출간지난 8월 조영호 교수님의 저서 『이것이 이공계다』가 출간되었다. 조영호 교수님은 UC 버클리(Univ. of Ca...
2018-10-16 -
 [졸업생취재] 정인경 교수(현, KAIST 생명과학과)
[졸업생취재] 정인경 교수(현, KAIST 생명과학과)정인경 교수님은 2006년 바이오시스템 (현 바이오및뇌공학) 에서 B.S, 2011년 바이오및뇌공학 (PI 김동섭 교...
2018-10-16 -
 [연구실 대표 취재] 단백질 생물 정보학 연구실
[연구실 대표 취재] 단백질 생물 정보학 연구실8월 10일 금요일, 단백질 생물 정보학 연구실의 랩대표 손정태 학생과 인터뷰를 가졌습니다. Q. 연구실에 대...
2018-09-03 -
 [봄학기 CA 취재] 김효민(CA장)과 김준희 조교
[봄학기 CA 취재] 김효민(CA장)과 김준희 조교이번 학과 취재 인터뷰는 2018년 봄학기 동안 CA(학사과정 학업 및 진로 상담) 활동하셨던 김효민 CA장과 김...
2018-09-03 -
 [연구실 대표 취재] 바이오영상신호처리연구실
[연구실 대표 취재] 바이오영상신호처리연구실Q. 안녕하세요. Bio Imaging, Signal Processing & Learning(BISPL) LABORATORY에 대한 간단한 소개를 ...
2018-08-01 -
 [연구실 대표 취재] 생물정보학 및 합성생물학 연구실
[연구실 대표 취재] 생물정보학 및 합성생물학 연구실Q. 연구실에 대해 간단히 소개 해주실 수 있을까요? 저희 연구실은 이관수 교수님의 지도 하에 분자 수준에...
2018-08-01 -
 [연구실 대표 취재] 바이오정보시스템 연구실
[연구실 대표 취재] 바이오정보시스템 연구실Q. 안녕하세요. BIO-INFORMATION SYSTEM LABORATORY에 대한 간단한 소개를 부탁드려도 될까요? 저희 연구실...
2018-07-03 -
 [연구실 대표 취재] 시스템생물학 및 바이오영감공학 ...
[연구실 대표 취재] 시스템생물학 및 바이오영감공학 ...6월 4일 월요일, 시스템생물학 및 바이오영감공학 연구실의 랩대표 강의룡 학생과 인터뷰를 가졌습니다. Q. ...
2018-06-04 -
 [연구실 대표 취재] 나노감응시스템연구실
[연구실 대표 취재] 나노감응시스템연구실Q. 안녕하세요. 나노감응시스템 연구실 (NanoSentuating System Laboratory)에 대한 간단한 소개를 부탁드려...
2018-05-30 -
 [연구실 대표 취재] 오믹스연구실
[연구실 대표 취재] 오믹스연구실화창한 5월 3일 목요일, OMICS 연구실의 박사과정 방효은 (랩대표), 장기원 학생과 인터뷰를 가졌습니다. Q....
2018-05-03 -
 [연구실 대표 취재] 바이오인터페이스전자소자 연구실
[연구실 대표 취재] 바이오인터페이스전자소자 연구실Q. 안녕하세요. 바이오인터페이스 전자소자(BIE) 연구실에 대한 간단한 소개를 부탁드려도 될까요? 현대 시...
2018-04-30 -
 [연구실 대표 취재] 신경공학연구실
[연구실 대표 취재] 신경공학연구실4월 19일 목요일, 신경공학 연구실의 랩대표인 박사과정 김대정 학생과의 인터뷰를 가졌습니다. Q. 연구실에...
2018-04-19 -
 [연구실 대표 취재] 인지신경영상 연구실
[연구실 대표 취재] 인지신경영상 연구실3월 21일 수요일, 인지신경영상 연구실 랩대표인 정진용 학생과의 인터뷰를 가졌습니다. 정진용 학생은 KAIS...
2018-04-03 -
 [연구실 대표 취재] 자기공명영상 연구실
[연구실 대표 취재] 자기공명영상 연구실Q. 안녕하세요. 자기공명영상(MRI) 연구실에 대한 간단한 소개를 부탁드려도 될까요? 저희 연구실에서는 인...
2018-04-03 -
 [연구실 대표 취재] 시각신경시스템 연구실
[연구실 대표 취재] 시각신경시스템 연구실인터뷰는 2월 19일 월요일, 양분순 빌딩 3층 복도에서 진행되었다. 최우철 석박사통합과정 학생은 카이스트 ...
2018-03-20 -
 [연구실 대표 취재] 계산 신경 생리학 연구실
[연구실 대표 취재] 계산 신경 생리학 연구실Q. 간단하게 연구실 소개 부탁 드립니다. A. 우리 연구실의 목표는 뇌 기능의 일반원리 (the general princi...
2018-03-20 -
 [연구실 대표 취재] 바이오포토닉스 연구실
[연구실 대표 취재] 바이오포토닉스 연구실Q. 연구실 소개를 간단하게 부탁 드립니다. A. 저희는 의광학기술을 바탕으로 내시현미경 (Micro-endoscopy)...
2018-03-20 -
 [연구실 대표취재] 나노바이오공학연구실
[연구실 대표취재] 나노바이오공학연구실Q. 연구실 소개를 간단하게 부탁 드립니다. A. 저희 연구실은 박제균 교수님의 지도 아래 2018년 기준으로 ...
2018-01-31 -
 [연구실 대표 취재] 뇌기계지능 연구실
[연구실 대표 취재] 뇌기계지능 연구실인터뷰는 1월 26일 금요일, 뇌기계지능 연구실 (양분순 401호)에서 진행되었다. 컴퓨터 앞에서 연구에 열중...
2018-01-31 -
 [연구실 대표 취재] Memory and Cognition 연구실
[연구실 대표 취재] Memory and Cognition 연구실Interviewer: 이근민 Interviewee: 강준영 간단한 연구실 소개 및 설명 부탁드립니다. 우리 연구실은 기억에...
2018-01-31 -
 [연구실 대표 취재] 신경물리학 연구실
[연구실 대표 취재] 신경물리학 연구실[연구실 대표 취재] 신경물리학 연구실 – 이정민 박사과정 학생 인터뷰는 12월 28일 목요일, 신경물리학 연...
2017-12-31 -
 [학과 교수 취재] 박성홍 교수
[학과 교수 취재] 박성홍 교수자기공명영상 연구실 박성홍 교수님. 20년간 자기공명영상 기술을 연구하시며, 현재 해당 분야를 선도하고 ...
2017-12-31 -
 [연구실 대표 취재] 세포신호및생체영상 연구실
[연구실 대표 취재] 세포신호및생체영상 연구실세포신호및생체영상 연구실 Interviewer: 이근민 Interviewee: 최호준 연구실에 대한 간단한 소개 부탁드립...
2017-12-31 -
 [연구실 대표 취재] 생체재료공학연구실
[연구실 대표 취재] 생체재료공학연구실Q. 연구실 소개를 간단하게 부탁 드립니다. A. 저희 연구실은 생체재료공학 연구실(Biomaterials Engineerin...
2017-11-30 -
 [학과 교수 취재] 피오릴로 교수 (Prof. Fiorillo)
[학과 교수 취재] 피오릴로 교수 (Prof. Fiorillo)Interviewee: Prof. Fiorillo Interviewer: 이근민 (KeunMin Ken LEE) What is your current research about...
2017-11-30 -
 [학과 교수 취재] 예종철 교수
[학과 교수 취재] 예종철 교수KAIST 바이오및뇌공학과 교수님을 취재하다 바이오영상신호처리 연구실 예종철 교수님 인터뷰는 11월 16일 ...
2017-11-30 -
 [연구실 대표 취재] 생물물리학/바이오나노구조 연구실
[연구실 대표 취재] 생물물리학/바이오나노구조 연구실간단한 연구실 소개 부탁드립니다. 어떤 연구를 하고 있나요? 안녕하세요, 바이오및뇌공학과 생물물리학/바...
2017-10-30 -
 [연구실 대표 취재] 생체미세환경 연구실
[연구실 대표 취재] 생체미세환경 연구실[연구실 대표 취재] 김효민 박사과정 – 생체미세환경 연구실(BIMIL) 우리 과의 김필남 교수님께서 이끌고 계...
2017-10-30 -
 [학과 교수 취재] 이수현 교수
[학과 교수 취재] 이수현 교수KAIST 바이오및뇌공학과 교수님을 취재하다 - 기억인지연구실 이수현 교수님 인터뷰는 2017년 10월 30일, 이...
2017-10-30 -
 [학과 교수 취재] 박지호 교수
[학과 교수 취재] 박지호 교수[학과 교수 취재] 박지호 교수 박지호 교수님은 미국 University of California, San Diego에서 재료공학과...
2017-09-30 -
 [학과 교수 취재] 이상아 교수
[학과 교수 취재] 이상아 교수KAIST 바이오및뇌공학과 교수님을 취재하다 인지발달연구실 이상아 교수님 인터뷰는 9월 27일 화요일, 카이...
2017-09-30 -
 [학과 교수 취재] 조영호 교수
[학과 교수 취재] 조영호 교수2017년 봄학기 학과연구성과취재 조영호 교수님 Interviewee: 조영호 교수님 Interviewer: 한윤호 조교 Q1. ...
2017-09-30 -
 2017 봄학기 학과 성과 취재 조교 활동을 마치며
2017 봄학기 학과 성과 취재 조교 활동을 마치며취재 조교: 김래영, 송영조, 한윤호 2017년 봄학기 학과 성과 취재 조교 활동이 마무리되었습니다. 활동이 ...
2017-08-31 -
 [학과 교수 취재] 이도헌 교수. 모든 문제는 해법이 있...
[학과 교수 취재] 이도헌 교수. 모든 문제는 해법이 있...바이오정보시스템 연구실 이도헌 교수님 어떤 계기로 바이오정보학(bioinformatics) 분야를 연구하게 되셨나...
2017-07-30 -
 [학과 교수 취재] 박제균 교수
[학과 교수 취재] 박제균 교수Interviewee: 박제균 교수님 Interviewer: 한윤호 조교 Q1. 교수님께서는 2002년부터 카이스트에서 교수직을...
2017-07-30 -
 [학과 교수 취재] 남윤기 교수
[학과 교수 취재] 남윤기 교수- 신경 공학 연구실, 남윤기 교수님 인터뷰는 7월 25일 화요일, 교수님의 사무실에 진행되었다. 평소에 학과...
2017-07-30 -
 [학과 교수 취재] 김필남 교수
[학과 교수 취재] 김필남 교수Interviewee: 김필남 교수님 Interviewer: 한윤호 조교 Q1. 교수님께서는 서울대학교에서 대학원 및 포닥(박...
2017-06-30 -
 [학과 교수 취재] 김동섭 교수. 할 수 있는 일에 최선...
[학과 교수 취재] 김동섭 교수. 할 수 있는 일에 최선...생물정보학 및 전산생물학 연구실 김동섭 교수님 교수님의 연구 분야에 관해 설명해 주세요. 어떤 연구를 하...
2017-06-30 -
 [학과 교수 취재] 정용 교수
[학과 교수 취재] 정용 교수- 인지신경영상 연구실, 정용 교수님 인터뷰는 5월 16일 화요일, 교수님의 사무실에 진행되었다. 사무실 위...
2017-06-30 -
 [학과 교수 취재] 이관수 교수
[학과 교수 취재] 이관수 교수학부 전공이 생물학인데 어떻게 생물정보학 연구를 하게 되셨나요? 학부 때 생물학을 전공했지만 스스로 화...
2017-05-30 -
 [학과 교수 취재] 최철희 교수
[학과 교수 취재] 최철희 교수Interviewee: 최철희 교수님 Q1. 교수님께서는 세포 신호전달, 생체의학 이미징, 바이오광학, 엑소좀 등 다...
2017-05-30 -
 [학과 교수 취재] 이상완 교수
[학과 교수 취재] 이상완 교수- 뇌기계지능 연구실, 이상완 교수님 인터뷰는 5월 15일 월요일, 교수님의 사무실에 진행되었다. 사무실에 ...
2017-05-30 -
 [학과 교수 취재] 백세범 교수
[학과 교수 취재] 백세범 교수- 시각신경시스템 연구실, 백세범 교수님 인터뷰는 4월 21일 금요일, 교수님의 사무실에서 진행되었다. 교수...
2017-04-30 -
 [학과 교수 취재] 최정균 교수
[학과 교수 취재] 최정균 교수1. 교수님께서는 KAIST에서 학부와 대학원 과정을 모두 마치시고 교수로 임용되셨습니다. 학교 생활을 하실 ...
2017-04-30 -
 [학과 교수 취재] 조광현 교수. 자신만의 질문과 그 대...
[학과 교수 취재] 조광현 교수. 자신만의 질문과 그 대...시스템생물학 및 바이오영감공학 연구실 조광현 교수님 인터뷰 어떤 계기로 시스템생물학을 연구하게 되셨나...
2017-04-30 -
 빛과 전기로 세포를 움직이는 법 새로운 광전자 유체 ...
빛과 전기로 세포를 움직이는 법 새로운 광전자 유체 ...우리 학과 박제균 교수 연구팀이 세포와 마이크로 입자의 패터닝이 가능한 광전자적 유체 프린팅 시스템을 ...
2017-03-30 -
 인공 신경망을 이용한 MRI 이미지 결함 수정법 개발
인공 신경망을 이용한 MRI 이미지 결함 수정법 개발박성홍 박사 연구팀은 인공신경망 기법은 MRI 데이터에 적용해 MRI 이미지 결함을 수정하는 기술을 개발했다...
2017-03-30 -
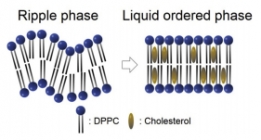 X-ray를 이용하여 세포막간과 콜레스테롤 농도의 관련...
X-ray를 이용하여 세포막간과 콜레스테롤 농도의 관련...세포의 세포막에는 여러 구성성분이 있지만 그 중에서도 콜레스테롤은 세포막의 유동성을 조절하는데 중요한...
2017-03-30 -
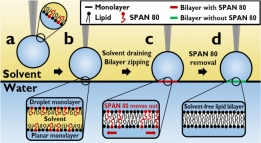 안정적인 대면적의 세포막 모델을 구축할 수 있는 시스...
안정적인 대면적의 세포막 모델을 구축할 수 있는 시스...세포의 세포막은 여러 종류의 단백질과 탄수화물을 비롯하여 주성분은 인지질로 이루어져 있다. 이 인지질은...
2017-01-17 -
 초고민감 실리콘 나노 박막의 유전체 메타물질 개발
초고민감 실리콘 나노 박막의 유전체 메타물질 개발지난 11월 우리 학과 정기훈 교수 연구팀은 미세한 양의 생화학 물질을 검출할 수 있는 유전체 메타물질을 ...
2017-01-17 -
 인간의 뇌를 모방한 음성 인식 기술 개발
인간의 뇌를 모방한 음성 인식 기술 개발지난 11월 우리학과 조광현 교수 연구팀은 뇌가 음성을 인지하는 메커니즘을 모방하여 노이즈 대비 신호 분...
2017-01-17 -
 3차원 DNA 구조를 통한 중요 발암인자 발굴 및 인공지...
3차원 DNA 구조를 통한 중요 발암인자 발굴 및 인공지...암의 원인이 되는 ‘driver mutation’을 찾는 것은 암 유전체학에서 가장 중요하며 어려운 문제 중 하나이다....
2016-12-19 -
 알츠하이머 병과 관련된 타우 단백질의 역할 메커니즘 ...
알츠하이머 병과 관련된 타우 단백질의 역할 메커니즘 ...연구 분야의 트렌드 2015년 보건복지부 자료에 따르면, 2020년에는 전체 노인 인구 중 치매환자의 비중이 10...
2016-12-19 -
 생체의 안전벨트 역할을 해낼 수 있을까, 더블 네거티...
생체의 안전벨트 역할을 해낼 수 있을까, 더블 네거티...지난 9월, 우리학과 조광현 교수 연구팀은 체내에서 발생하는 신호의 빠르기를 탐지하는 신호전달물질 네트...
2016-12-19 -
 고감도 화학물질 검출을 위한 사용자 친화적 툴 개발
고감도 화학물질 검출을 위한 사용자 친화적 툴 개발지난 10월, 우리학과 박제균 교수 연구팀은 기존 상용화에 어러움이 있었던 고감도 화학물질 검출 시스템을 ...
2016-12-19 -
 혈중 암세포 진단을 위한 양면형 진단 장치의 개발
혈중 암세포 진단을 위한 양면형 진단 장치의 개발지난 10월 우리학과 조영호 교수 연구팀은 혈액 속에 존재하는 암세포를 진단하는데 필요한 양면형 진단 장...
2016-12-19 -
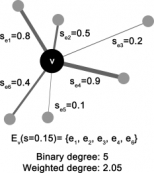 뇌 영상 분석 위한 새로운 통계 분석법 개발
뇌 영상 분석 위한 새로운 통계 분석법 개발지난 9월 우리학과 정용 교수 연구팀은 뇌 영상 신호 분석을 위한 새로운 통계 분석법을 발표했다. 본 연구 ...
2016-12-18